小社が立地する熊本県の生命科学研究最前線を、熊本大学の若手研究者が連載(8回)でお届けします。
RNA 高次構造と遺伝子発現制御との関連性について
Causative Links between RNA Higher-Order Structures and Controlling Gene Expression

五木 結愛
熊本大学大学院
先端科学研究部
博士後期課程

勝田 陽介
熊本大学大学院
先端科学研究部
准教授
Abstract Recent advancements in scientific technology have revealed that RNA, forms complex higher-order structures that play crucial roles within cells. This review focuses on the relationship between RNA's higher-order structures and gene expression, highlighting technological advancements and specific functions.
1. はじめに
我々は高校生物の教科書で「二重螺旋構造の DNA」から転写反応が進行することで「紐状の RNA」が作製され、翻訳反応という過程を踏んで「複雑な構造を持つタンパク質」が出来上がるというプロセスを学習してきた。
複雑な構造を形成しているタンパク質は、熱変性などによって構造が崩れると失活することからもわかる通り、その機能において構造が重要な役割を担っていることが以前から知られていた。ところが近年の科学技術の進歩に伴い、核酸も同様に複雑な高次構造を形成しており、この高次構造が細胞内において重要な役割を果たしていることが明らかになってきている。
例えばほとんどの領域が相補鎖と二重螺旋構造を形成していると考えられていた DNA に関しては、ゲノム DNA 末端の一本鎖領域でグアニン四重鎖(G4)構造を形成していることが報告されており、神経変性疾患などと密接に関連している可能性が見出されている 1)。こうした事実は、相補鎖が存在しない核酸の領域が分子内構造を形成しやすいことを示唆しているとも言える。
つまり、相補鎖が存在しないため一本鎖の状態であると考えられていた RNA は、分子内の塩基対形成などによって DNA と比較してはるかに多様な分子内高次構造を形成していると考えられる。実際に、熱力学的安定性に基づく二次構造予測や構造解析シーケンス技術などによって RNA の高次構造が解き明かされるにしたがって、遺伝子発現など多くの生体内における反応と RNA の構造が密接に関連していることが見出されつつある。
本レビューでは、こうした RNA の複雑な構造と遺伝子発現の関連性についてまとめる。
2.「構造」解析技術の発展
RNA の高次構造は、かねてより酵素マッピング法といった生化学的な手法で解析されてきた。この手法は、エンドヌクレアーゼとエキソヌクレアーゼを組み合わせて RNA の一本鎖領域を特定し標的 RNA の全体構造を解明するものである。こうした手法によって塩基配列という一次構造ではなく二次構造が重要な意味を持つハンマーヘッドリボザイムといった「機能的な RNA」の存在が見出され、配列と構造の関連を探る研究の必要性に大きな注目が集まったといえる 2)。つまり lncRNA のようなタンパク質に翻訳されない RNA は高次構造形成によって生体内で重要な役割を担っており、タンパク質に翻訳される mRNA も単なる遺伝情報の中継ぎではなく高次構造形成によって遺伝子発現を制御している可能性が示唆されるようになり、高次構造解析技術の必要性が認識されるようになったということだ。しかしこうした手法のみで数十万~数百万種存在すると言われている RNA 全ての高次構造を解き明かすことは非現実的であり、大規模解析を可能とする手法の開発が必要となる。こうした背景から次世代シーケンサー(NGS)といったハイスループットな技術を用いることで網羅的に構造解析を行う研究が進められ、数多くの RNA に対する生化学的な手法での構造解析が可能になってきた 3-5)。
また、計算科学に基づく高次構造予測技術も近年著しい発展を遂げている。以前より熱力学的安定性に基づいてRNA の平面構造を予測するツールとして mRNAfold や RNAStructure などが広く用いられてきた 6)。しかしこれらのツールで予測できるのは局所的な配列情報から算出された構造に限定されている。そこで RNA 全体がとりうる構造を包括的かつ正確に予測するために、AI が活用され始めた。機械学習・深層学習により局所的な配列情報だけでなく RNA 全体の構造を考慮できるようになり、長鎖の RNA に対する計算科学的な手法での構造予測が可能になってきた 7)。
さらに上記の実験や計算に基づいて予測された RNA の高次構造について直接的に観測し、構造を決定する技術も大きく進歩している。以前から、NMR や X 線構造解析技術が RNA の高次構造を解析するために用いられてきた。しかしこれらの技術では RNA の局所的な高次構造やタンパク質との部分的な相互作用を解明するにとどまり、RNA 全体がどのよ うな高次構造をとるかについての情報を取得することは困難であった。こうした背景から cryo EM といった RNA 全体の構造を捉える技術の研究が進められてきた。 RNA 全体の構造に対する直接的な情報が得られるようになったことで、長鎖の RNA に対する直接的な構造解明が可能になってきた 8)。
上記のような技術の発展により 、RNA の高次構造解析が効率的かつ高精度に行われるようになった。これにより多くの RNA 配列の構造が解明され、その構造が持つ機能が次々と明らかにされている。
3.UTR で形成される高次構造の機能
ヒトを代表とする高度な生命体において、リボソームは mRNA の最上流つまり 5’UTR のキャップ構造にリクルートされ、5’UTR をスキャンし開始コドンからアミノ酸伸長反応を開始する。しかし、ウイルスや細菌といった単純な遺伝子しか持たない生命体(ウイルスを生命と判定するか否かは別として)は、可能な限り多様なタンパク質の発現を可能とするシステムが必要である。この鍵となっている RNA 高次構造が「Internal Ribosome Entry Site: IRES」である。
これは複数のステムループ等から成る複雑な高次構造であり、キャップ構造非依存的にリボソームを mRNA へ直接リクルートする。イメージしやすいように説明するとすれば高速道路のインターチェンジから車が侵入するようなもので、リボソームが 5’UTR の末端であるキャップ構造からではなく、 mRNA のいわゆる「端っこではない」領域である IRES からエントリーし下流の遺伝子配列の翻訳が開始される。
IRES はこれまでにウイルスをはじめ様々な生物のゲノムから 100 種類以上が同定されており、高次構造や翻訳開始の作用機序によって 4 つのクラスに分類できることが知られている。ClassⅠに分類される IRES はピコルナウイルス科によく見られ、複数のステムループ構造からなるものの、Class Ⅰ から Class Ⅳに分類される IRES の中では最もシンプルな構造を形成している 9-11)。IRES としての作用機序は eIF4G などの翻訳開始因子と結合することでリボソームのリクルートを促すドメインを有し、一般的な mRNA と同様にリボソームが翻訳を開始する。Class Ⅱに分類される IRES は、カルジオウイルス属やアフトウイルス属に多く見られ、Class Ⅰと同様にシンプルな高次構造から成る 12, 13)。この Class に分類される IRES は eIF4G 結合ドメイン直下に開始コドンが存在することから、eIF4G にリクルートされたリボソームは開始コドンを探索する必要なく翻訳を開始することができる特徴がある。つまり Class Ⅰと Class Ⅱは構造そのものはシンプルであるものの、開始コドンの探索が必要であるか否かで区別化されている。 Class Ⅲに分類される IRES はフラビウイルス科に多くみられ、ステムループ構造に加えてシュードノット構造などによる複雑な構造を形成している 14, 15)。 eIF3 およびリボソームの小サブユニットは Class Ⅲの IRES 上で結合することから、リボソームは IRES 直下の開始コドンからタンパク質翻訳が開始できる 。Class Ⅳに分類される IRES はジシストロウイルス科から多数が見つかり、Class Ⅲと同様にシュードノット構造を含む複雑な高次構造を形成している 16-18)。この Class に分類される IRES は mRNA の翻訳領域上流に存在するのではなく、2 つの翻訳領域の間を繋ぐ IGR(Intergenic Region)に存在し、IRES 下流に位置する遺伝子のタンパク質の翻訳を可能とする。機序としては、Class Ⅳ に分類される IRES がリボソームの小サブユニットと大サブユニットの両方をリクルートし、非開始コドンである IRES 直下の GCU(Ala)からの翻訳を促す。
先述のような IRES のケースを除外するとすれば、一般的にリボソームは mRNA の最上流つまり 5’UTR のキャップ構造から侵入し、開始コドンから翻訳を開始する。つまり、このプロセスに影響を与える「何か」はもちろんタンパク質の翻訳反応に影響を与えるだろう。例えば、オルニチン脱炭素酵素 ODC の 5’UTR は GC 含有率が高いため安定した、いわゆる硬いステムループ構造を形成しており、これらがリボソームのスキャニングを阻害することでタンパク質翻訳を抑制する 19)。また鉄輸送関連遺伝子フェリチンの 5’UTR に存在する鉄応答エレメント(IRE)と呼ばれる構造は、IRP というタンパク質と結合しリボソームが翻訳開始複合体へ結合するのを立体障害によって阻害するため、タンパク質翻訳は抑制される 20)。
上記した「何か」次第では、タンパク質発現量が減るのみではなく増えるという現象につながることもある。例えばインスリン様成長因子 IGF-1 の 5’UTR のステムループは Poly A Binding Protein(PABP)というタンパク質と結合し、 mRNA 上流における翻訳開始複合体の形成を促すことでタンパク質翻訳を促進するケースなどが知られている 21, 22)。
一方で、 mRNA にはリボソームによるタンパク質翻訳反応が終結した後の領域、つまり 3’UTR が存在しており mRNA の安定性と関係していると言われている。もちろんこの領域においても、遺伝子発現に影響を与える「何か」の例は数多く報告されている。例えば、一時的に発現することが重要であることがわかっている免疫関連遺伝子インターロイキン 6 の 3’UTR に存在するステムループ構造が RNA 分解酵素 Rignase-1 と結合し、 mRNA そのものの分解速度を上げることでタンパク質翻訳を抑制する 23)。また同じく免疫関連遺伝子 TNFα の 3’UTR には A や U に富んだ領域が存在し(AU リッチエレメント:ARE)、この領域で形成される RNA 高次構造が mRNA 分解酵素 Tristetraprolin(TTP)と結合することでタンパク質翻訳を抑制する 24-26)。
またこの 3’UTR においても「何か」次第ではタンパク質発現量が増える。例えば、試験管レベルおよび細胞レベルでの検討ではあるものの、標的 RNA の 3’UTR に GC リッチな安定高次構造などが存在するとエキソヌクレアーゼの進行が阻害され、結果として RNA の安定性が向上することでタンパク質翻訳量は増加する報告が挙げられている 27)。この事実から察するに、内在性の遺伝子においても、同様の機序で mRNA を安定化し遺伝子発現量を増やす高次構造が存在する可能性は大きいであろう。
このようにステムループ等から構成される「機能的な RNA」の高次構造は、遺伝子配列の数だけ無数に存在しており、形成位置や配列によって非常に多様な効果を示す。そのため、前項の構造解析技術や構造予測技術を駆使して、配列ごとに異なる RNA の高次構造や RNA 結合タンパク質との相互作用を解析することが重要になる。
4.RNA G4 と遺伝子発現の関連
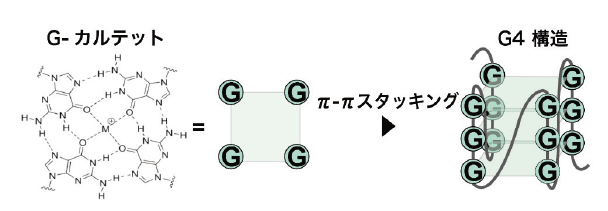
RNA G4 構造は 4 つのグアニン塩基がフーグスティーン型塩基対によって平面構造(G-quartet)を形成し、これらが π スタッキングにより積み重なり強固な高次構造を形成する(図 1)。ステムループ構造は配列の数だけ多様な種類が存在するのに対し、 RNA G4 はグアニンの連続配列という単純な要素で形成される構造であるにもかかわらず、その存在する「場所」によって遺伝子発現を増減させる機能の多様性に注目が集まっている。
前項で記載した通り、リボソームのスキャニングに強い影響を与える 5’UTR に高次構造が存在するとタンパク質の発現が抑制される。つまり RNA G4 のような強固な高次構造が 5’UTR に存在すれば、タンパク質発現を強く抑制することは容易に想像できるだろう。例えばがん関連遺伝子 NRAS や Zic-1 などがその例として知られる28, 29)。
また 5’UTR に存在する RNA G4 も遺伝子によっては、タンパク質発現量が増えることもある。例えばアポトーシス阻害遺伝子 clAP1 の 5’UTR に存在する RNA G4 は、詳細な機構については明らかになっていないもののタンパク質翻訳量を増やす効果があることが報告されている 30)。
一方で 3’UTR に存在する RNA G4 に関しても遺伝子発現に影響を与える例が数多く報告されている。例えば、アミロイド前駆体タンパク質 APP やリン酸化酵素 PIM1 の 3’UTR に存在する RNA G4 は多くの RNA 結合タンパク質と相互作用しタンパク質翻訳を抑制することが報告されている 31, 32)。しかしそれ以上の詳細なメカニズムに関しては未だ明らかになっていない。
また、3’UTR に存在する RNA G4 も遺伝子によってはタンパク質翻訳量を増やすことがある。脂肪酸不飽和化酵素遺伝子 desaturase の 3’UTR に存在する RNA G4 は、 mRNA そのものの分解を誘導する miRNA(micro-RNA)の一種である mir331-3p の結合領域上に存在しており、標的 mRNA への mir331-3p の結合を阻害することで mRNA の分解耐性を向上させ、結果としてタンパク質翻訳量を増加させる効果がある 33)。
そのほかの遺伝子についても網羅的に解析を行った結果 miRNA の結合領域と重複する RNA G4 形成配列が 44,000 箇所同定されており、 RNA G4 が標的 mRNA への miRNA の結合を阻害することで mRNA の分解を抑制している可能性が示唆される。さらに、低密度リポタンパク質受容体関連タンパク質 LRP5 の 3’UTR に存在する RNA G4 は代替ポリアデニル化を促進し、 miRNA の結合領域を含まない短い 3’UTR を生成 することで、 mRNA の分解を抑制しタンパク質発現量を増加させることも報告されている 34)。
上記においては mRNA、つまり遺伝子の情報を「持つ」 RNAについて言及してきた。これに加え、近年、遺伝子の情報を「持たない」 RNA も、 RNA G4 などの高次構造を介して遺伝子発現に影響を与えることがわかってきた。また、その存在比に関しては RNA 全体のうち遺伝子の情報を「持つ」 RNA はわずか 2%で、遺伝子の情報を「持たない」 RNA は 98% 存在することになる。この 98%と生体内の “ほぼ” 全てを占める遺伝子の情報を持たない RNA は ncRNA(non-coding RNA)と呼ばれている。かつて ncRNA は「ジャンク RNA」だと考えられていたが、その多くが高次構造の形成によって多くの機能を有する「機能的な RNA」であることが明らかとなっている。
様々な種類が存在する ncRNA は長さによって大きく 2 つに分けられる。500 nt 以上に及ぶ長鎖の ncRNA は lncRNA(long non-coding RNA)と呼ばれ、MALAT1 や NEAT1 など核内において高いレベルで発現していることが知られている。例えば、MALAT1 に存在する RNA G4 は NONO というタンパク質と結合し、核内における MALAT1 の局在化を誘導することでがん関連のタンパク質翻訳は抑制される 35)。
また長さが 20 nt 程度の非常に短い ncRNA のうち、遺伝子サイレンシング機能を有する miRNA にも RNA G4 は数多く存在している。 miRNA は通常 AGO タンパク質と RISC 複合体を形成し、相補的な配列を持つ mRNA に結合することで標的 mRNA を分解し遺伝子発現を抑制する。つまり、この miRNA 中に存在する RNA G4 構造は、 miRNA とAGO タンパク質の複合体形成を妨げ、 miRNA と標的 mRNA の結合を阻害し遺伝子発現抑制機能を失わせる。
すでに、 RNA G4 の構造基盤となる G-quartet の存在が明らかになってから半世紀以上が経過している。しかし近年の計算化学や構造解析技術の進歩に伴い、 RNA G4 の形成位置や機能についての知見が増えているものの、未だその全貌は解き明かされていない。したがって RNA G4 の機能を明らかにするために、日々進化する分析技術を駆使して RNA G4 の位置やタンパク質との相互作用を正確に把握していくことが求められる。
5.おわりに
本総説では、 RNA G4 をはじめとする RNA 上の高次構造が遺伝子発現を減らす・増やす例について述べた。このように近年では RNA が単なる遺伝情報の媒体物質ではなく、高次構造によって様々な機能を有する「機能的な RNA」であり、ストレス応答、シグナル伝達など、多岐にわたる生理機能に関与していることが多くの研究によって示されている。今後の研究は、これらの知見を基に RNA の機能をより深く理解し、応用するための新しい方法を模索することが求められるであろう。具体的には、計算化学や構造解析技術の更なる進化などが挙げられる。
最後に、「機能的な RNA」の高次構造やその機能を明らかにすることは、基礎研究のみならず医療やバイオテクノロジーなどの分野においても大きく貢献できる可能性を秘めている。今後の「機能的な RNA」に関する研究が、多くの分野においてこれまでにない新しい発見と応用をもたらすであろう。
【参考文献】
- E. Renton et al., Neuron, 2011, 72(2), 257-268.
- C. Anthony et al., Cell, 1987, 49, 211-220.
- C. Cao et al., Nat. Commun., 2021, 12(1), 3917.
- S. Rouskin et al., Nature, 2014, 505, 701-705.
- Q. Luo et al., Nat. Commun., 2021, 12, 3397.
- J. S. Reuter et al., BMC Bioinformatics, 2010, 11, 129.
- K. Sato et al., Nat. Commun., 2021, 12, 941.
- S. Imai et al., Nat. Commun., 2023, 14, 4977.
- E. Anderson et al., J. Gen. Virol., 2007, 88, 3043-3052.
- J. M. Bailey et al., J. Virol., 2007, 81, 650-668.
- J. Lin et al., Nucleic. Acids Res., 2009, 37, 47-59.
- G. Lozano et al., Curr. Opin. Virol., 2015, 12, 113-120.
- Y. Gao et al., Virol. J., 2016, 13, 107.
- M. Asnani et al., Virology, 2015, 478, 61-74.
- G. Belsham, Virus Res., 2009, 139, 183-192.
- C. H. Kerr et al., J. Virol., 2016, 90, 5538-5540.
- E. Jan et al., J. Mol. Biol., 2002, 324, 889-902.
- T. V. Pestova et al., Genes Dev., 2007, 17, 181-186.
- J. Manzella et al., J. Biol. Chem., 1990, 265, 20.
- M. Martina et al., Molecular Cell, 1998, 2, 383-388.
- O. P. De Melo Neto et al., Nucleic. Acids Res., 1995, 23, 2198-2205.
- S. D. Kulkarni et al., J. Biol. Chem., 2011, 286, 14146-14156.
- H. Iwasaki et al., Nat. Immunol., 2011, 12, 1167-1175.
- P. Anderson, Ann. Rheum., 2000, 59, i3-i5.
- A. B. Shyu et al., Cell, 2000, 102, 135-138.
- C. J. Wilusz et al., Nat. Rev. Mol. Cell. Biol., 2001, 2, 237-246.
- V. Solodushko et al., Gene Ther., 2023, 30, 620-627.
- S. Kumari et al., Nat. Chem. Biol., 2007, 3, 218-221.
- A. Arora et al., RNA, 2008, 14(7), 1290-1296.
- A. Singha Roy et al., Biochemistry, 2024, 63(4), 475-486.
- E. Crenshaw et al., PLoS ONE, 2015, 10(11), e0143160.
- A. Arora et al., RNA Biology, 2011, 8(5), 802-805.
- S. Rouleau et al., RNA, 2007, 23, 1172-1179.
- J. D. Beaudoin et al., Nucleic. acids Res., 2013, 41, 5898-5911.
- X. Mou et al., Nucleic. Acids Res., 2022, 50(1), 397-410.
| [ 著者プロフィール ] | |
| 氏名 | 五木 結愛(Yua Itsuki) |
|---|---|
| 所属 |
熊本大学大学院先端科学研究部 博士後期課程 〒860-8555 熊本県熊本市中央区黒髪 2 丁目 39 番 1 号 TEL:080-9666-6740 |
| 出身学校 | 熊本大学 |
| 学位 | 現在 博士後期課程 1 年 |
| 専門分野 | 核酸化学、生物化学 |
| 現在の研究テーマ | 遺伝子発現を増やす核酸医薬の開発 |
| [ 著者プロフィール ] | |
| 氏名 | 勝田 陽介(Yousuke Katsuda) |
|---|---|
| 所属 |
熊本大学大学院先端科学研究部 〒860-8555 熊本県熊本市中央区黒髪 2 丁目 39 番 1 号 TEL:080-9666-6740 |
| 出身学校 | 京都大学大学院理学研究科 |
| 学位 | 博士(理学) |
| 専門分野 | 核酸化学、生物化学 |
| 現在の研究テーマ | 核酸医薬 |